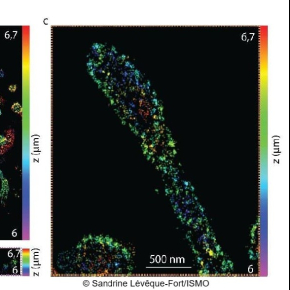
ModLoc localise des molécules en 3D avec une précision nanométrique
Des équipes de l’Institut des sciences moléculaires d’Orsay - ISMO (CNRS/Université Paris-Saclay) et de l’Institut Langevin1 ont mis au point une technique de microscopie en fluorescence qui atteint une précision nanométrique dans les trois dimensions.
Cet article est paru dans CNRS La lettre innovation de avril 2021, rubrique "Brevets et licences".
- 1CNRS/Sorbonne Université
Pour la recherche en biologie fondamentale, mais aussi en diagnostic médical, révéler la localisation en 3D de molécules dans des échantillons est un enjeu majeur. L'observation des cellules par microscopie en fluorescence, grâce aux techniques de super résolution, permet d'atteindre l'échelle du nanomètre. Mais avec les méthodes actuelles, les chercheurs butent encore sur un obstacle : pour mesurer la position axiale d'une molécule (dans l'épaisseur de l'échantillon), la précision varie avec la profondeur de l'observation.
Révéler la position tridimensionnelle de molécules avec des précisions nanométriques uniformes est aujourd’hui possible quelle que soit la profondeur à l’intérieur de l’échantillon biologique. Ces résultats de recherche originaux ont été obtenus par des équipes de l’ISMO et de l’Institut Langevin, en proposant un nouveau concept dans le processus de localisation des molécules uniques2 .
" Classiquement, l'échantillon est éclairé de manière uniforme. La nouvelle technique est fondée sur une illumination structurée variant rapidement au cours du temps ", explique Sandrine Lévêque-Fort, directrice de recherche CNRS à l’ISMO.
- 2En collaboration avec le laboratoire Physiopathogénèse et traitement des maladies du foie (Inserm/Université Paris-Saclay)
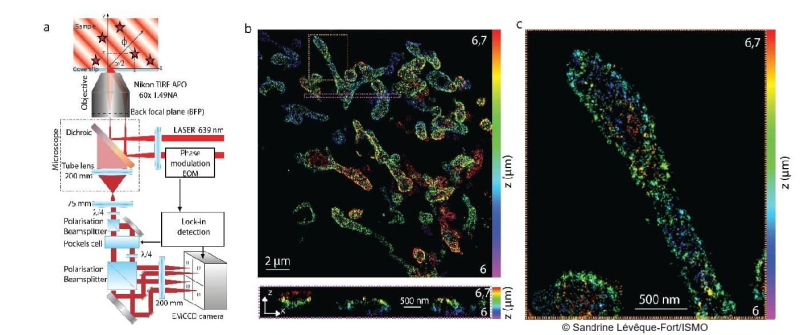
En effet, l'échantillon est éclairé par des franges sombres et lumineuses produites par l'interférence de deux faisceaux laser, franges qui se déplacent rapidement. L’émission de fluorescence des molécules est alors modulée temporellement, et révèle intrinsèquement la position de chacune des molécules. Cette technique appelée ModLoc (Modulated localization) permet d’atteindre des précisions axiales proches de la taille des biomolécules (6,8 nm). Il s’agit d’une réelle avancée pour les utilisateurs de la microscopie optique de super-résolution, qui cherchent à identifier précisément l’organisation en 3D des protéines au sein d'échantillons complexes. Avec la technique ModLoc, la résolution obtenue est uniforme jusqu’à plusieurs dizaines de microns de profondeur, et le champ d’observation reste compatible avec les applications biologiques visées.
ModLoc, qui a fait l'objet d'un dépôt de brevet, peut s’implémenter sur tout microscope optique. Le même principe, permettant d’extraire un paramètre de l’échantillon avec une précision constante à l’échelle nanométrique, pourrait s'appliquer à l’observation d’autres informations que la position, telle l'orientation des molécules. Une étude de valorisation est en cours, en lien avec la start-up Abbelight, spécialiste des technologies de microscopie en super-résolution, fondée suite à de précédents travaux issus des équipes de l’ISMO et de l’Institut Langevin.
Référence :
Nanometric axial localization of single fluorescent molecules with modulated excitation,
Pierre Jouchet, Clément Cabriel, Nicolas Bourg, Marion Bardou, Christian Poüs, Emmanuel Fort and Sandrine Lévêque-Fort.
Nature Photonics (janvier 2021)
Contact :
Sandrine Lévêque-Fort, Directrice de recherche CNRS à l'Institut des sciences moléculaires d’Orsay (ISMO), lauréate du Prix Irène Joliot-Curie 2020